RNA나 DNA 데이터들을 통계 처리까지 완료 했다면 그다음은 나온 후보 유전자들을 어떻게 해석할 것인가를 분석해야 한다.
해석하는 과정에 있어서 많이 사용하는 방법은 Gene Ontology와 Gene Set Enrichment Analysis 두가지이다.
그래서 이번편에서는 1. Gene Ontology와 GSEA가 무엇인지,
2. 두 방법의 차이점,
가능하다면 다음편에 3. Gene Ontology에 많이 사용되는 tool 'DAVID' 사용법을 공유하고자 한다.
- Gene Ontology란?
Gene Ontology는 예를 들어 RNA-seq 데이터로 DEG 분석을 시행하여 나온 관심 유전자들이 있을 것이다.
이 유전자들을 조사할 때 생물학의 어떤 pathway에 enrich되어 있고, 어떤 기능(function)을 하는지, 혹은 어떤 protein에 localized되어 있는지 annotation을 하기 위한 단계라고 보면 된다.
(어떤 pathway에 enrich가 되었음은 예를 들어 장에 염증이 나는 질병과 연관된 유전자들을 찾는 연구를 했다고 하자. 염증이 나는 이유는 면역력이 떨어져 생기는 경우들이 많으므로 후보군으로 나온 유전자들이 면역과 관련된 것들이 많은지 혹은 다른 기능의 유전자들이 많이 나왔는지 확인 할 수 있다)
♣Gene Ontology Categories(3)
1. Molecular Function: gene's jobs or abilities
2. Biological Process: events or pathways (ex-cell differentiation, development)
3. Cellular component: describe locations (ex: nucleus, cell membrane)
Gene ontology consortium
Gene ontology는 consortium에 의해 유전자들을 어떤 function을 해석할 것인지 3가지 카테고리로 나뉘어져 있다.그래서 DAVID tool에서도 3가지 카테고리 중에 어떤 것으로 해석하고 싶은지 선택하는 옵션에 있다.
- Gene Set Enrichment Analysis란?
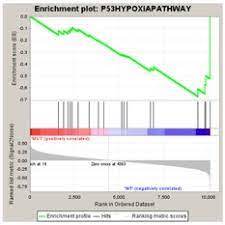
가끔 위에 같은 그림을 본 적이 있을 것이다. 이게 GSEA 결과로 나온 그림이다
- GSEA도 Functional enrichment analysis의 일종으로 유전자 후보군들(gene set)의 pathway를 파악하기 위해 하는 분석 방법이다.
- 같은 Gene pathway로 묶인 gene set들을 ChangeFold(DEG 결과에서 case와 control군 차이가 많이 나는 값)으로 가중치를 매겨서 얹어 -> ES "Enrichment score"를 매긴다.
- 이 Enrichment score를 통해 gene set들 중에서 중요한 유전자를 뽑아낼 수 있다는 장점이 있다.
GSEA가 gene set을 분석하는 방법은 다음과 같다.
- 모든 유전자들에 대해 ranking을 한다 -> fold change와 p-value로 가중치를 준다 -> 이 지정된 gene set에 포함되어 있는 모든 유전자들의 특정 영역에 몰릴 확률을 계산한다(Kolmogorov-Smirnov Statistic)
- Score summation, 모든 유전자들에 대해 합산한다
- 2번의 합산 최대치가 ES(Enrichment Score)을 확인해 gene set 내의 유전자 중 중요한 유전자군임을 추출할 수 있다.
- Permutation단계로, 샘플 phenotype을 permutation해 원래 데이터의 ES값을 비교해 p-value와 FDR를 계산한다.
- Gene Ontology와 GSEA의 차이점이란?
GO와의 차이점은 GO는 fisher test를 사용해 FDR로 cutoff쳐 자른다면 GSEA는 permutation과 rank를 사용한다는 점이다!
(방법의 차이와 더불어 GSEA는 gene set에서 중요한 유전자를 알 수 있다는 점, GO는 좀더 gene의 function에 대해 detail하게 살펴볼 수 있다는 장점이 있는 것 같다)
- GSEA 해석법
이번에는 그림을 보고 어떻게 해석하는지에 대해 공유하고자 한다.

사진 설명을 입력하세요.
위 그림에서 그림이 너무 여러개라 헷갈릴 수 있다ㅠ
차근차근 각각의 선을 먼저 설명하자면
먼저 중앙에 빨~파란선을 보자
빨간 색은 up-regulated gene, 파란색은 down-regulated gene을 의미하고
그리고 위에 있는 검정색 선은 같은 biological pathway를 가진 유전자들 set이다.
위에 초록선은 Enrichment Score을 의미한다.
맨 마지막 회색선은 가중치(weight)값을 말한다. (가중치는 logFold값에 의해 계산된 것이다!)
각각의 유전자들이 up/down regulation을 알 수 있으면서
Enrichment Score를 통해 어떤 유전자가 해당 biological pathway에서 중요한 역할을 하는지 알 수 있다.
이 그림에서 초록선이 가장 높은 곳에 있는 유전자가 up-regulated gene이면서 해당 생물학 pathway에서 중요한 역할을 하는 것을 알 수 있다.
나는 이 GSEA가 생소해서,,,이해하는데 한참걸렸던 것 같다...또 나만 그런거지..나만!!!!
다음편에 DAVID tool 사용법 쓴다고 했는데,,, 스크린샷 귀찮은데...할 수 있겠지 ...ㅠ
이상 주절주절...여기는 무시해주세요...ㅎㅎ
쓰고 싶은 건 많은데 시간이 없어서 화난다아아
